Le tendamistat vient se fixer au niveau du site actif par l'intermédiaire de trois acides aminés :
un tryptophane (18), une arginine (19) et une tyrosine (20). L'alpha-amylase est ainsi inhibée.
Nous pouvons voir que l'acarbose vient se fixer au niveau du site actif, inhibant ainsi l'enzyme.
b) Le tendamistat
Le tendamistat est une protéine synthétisée par une bactérie : Streptomyces tendae
Le gène codant pour l'alpha-amylase a subi une mutation qui a eu pour conséquence la modification du 197ème acide aminé de l'enzyme : l'acide aspartique a été remplacé par de l'alanine. Cette mutation rend l'alpha-amylase inactive.
3- Mise en évidence du mode d'action de deux inhibiteurs de l'alpha-amylase :
a) L'acarbose
L'acarbose est un pseudotétrasaccharide, d'origine microbienne. Au niveau de la bordure en brosse de l'intestin, l'acarbose agit par inhibition compétitive des alpha-glucosidases. Il diminue ainsi la dégradation des carbohydrates (dioligo- et polysaccharides) en monosaccharides absorbables.
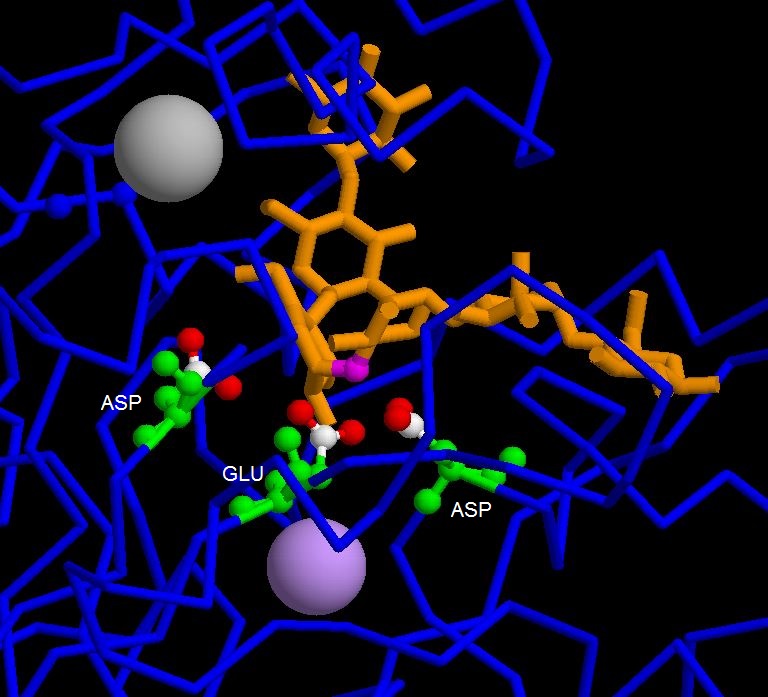
Le site actif de l'alpha-amylase est composé de 3 acides aminés : un acide glutamique (233) et deux acides aspartiques (197 et 300). Ce sont les groupements acide (en blanc et rouge) de ces acides aminés qui assurent principalement l'hydolyse de l'amidon.
Dans ce modèle, une courte chaîne de polysaccharide (en orange) est liée au site actif. Le site de coupure est coloré en rose.
Nous avons aussi fait apparaître deux ions : un ion calcium (en gris) qui stabilise la stucture de l'enzyme et un ion chlorure (en mauve) qui pourrait aider l'hydrolyse de l'amidon.
2- Mise en évidence du site actif de l'amylase "mutée" :
Protocole :
- A l'aide des fonctionnalités du logiciel Rastop, mettre en évidence et comparer le site actif de l'alpha-amylase "saine" et de l'alpha amylase "mutée".
- Mettre en évidence le mode d'action d'inhibiteurs sur l'alpha-amylase.
Matériel :
- Logiciel Rastop ou Rasmol
- Divers fichier PDB (format des molécules 3D) disponibles sur RCSB Protein Data Bank (http://www.rcsb.org/pdb/home/home.do)
Principe :
Lors de la formation du complexe enzyme-substrat, le substrat de l’enzyme vient se fixer au niveau du site actif de celle-ci. On appelle site actif la partie de l’enzyme impliquée directement dans l’intéraction enzyme-substrat.
L'amylase pancréatique est une enzyme libérée dans l’intestin impliquée dans la digestion de l’amidon. Son substrat est l’amidon ou des polysaccharides plus petits dont elle catalyse l’hydrolyse pour donner du maltose (diholoside) ou des dextrines (3 oses).
Certains individus ont une amylase "mutée" inactive ne parvenant pas à fixer ou à catalyser la transformation du substrat. On souhaite donner une explication moléculaire de cette inactivité et en retrouver l’origine.
A l'aide d'un logiciel de visualisation de molécules 3D, nous allons mettre en évidence le site actif de l'amylase ainsi que le complexe enzyme-substrat.